7 RNAfusion
8 Introduzione
Nonostante la bassa incidenza complessiva della rilevazione di geni di fusione nel cancro, numerosi approcci terapeutici hanno dimostrato un beneficio clinico in relazione al loro targeting specifico e alla modulazione della risposta immunitaria mediante terapie di precisione.
La presenza di geni di fusione mostra una correlazione significativa con l’inizio, la rilevazione e la progressione dei tumori. L’evidenza della rilevazione di tali segnali supporta l’ipotesi che essi rappresentino mutazioni “clonali”, ossia presenti in tutte le cellule neoplastiche della popolazione tumorale (review)[https://doi.org/10.1080/14796694.2025.2477974].

8.1 Arsenale di Strumenti a Disposizione
Esistono numerosi strumenti bioinformatici dedicati all’identificazione dei geni di fusione. Chimerascan rappresenta uno dei primi software sviluppati, caratterizzato da un’elevata sensibilità. Successivamente sono stati introdotti programmi come Defuse, EricScript e FusionCatcher, che ne hanno costituito un’evoluzione, migliorandone diversi aspetti in termini di accuratezza e prestazioni. In questo contesto verranno presentati alcuni strumenti singoli, di utilizzo relativamente semplice, insieme a un framework completo basato su Nextflow, che consente di integrarli e utilizzarli in maniera coordinata.
8.2 KALLISTO fusions
Kallisto può individuare le letture che attraversano potenziali punti di rottura di fusione (crossing-reads e split reads). La modalità quant aggiunge un’opzione –fusion che identifica le coppie di letture coinvolte in fusioni e scrive l’output nel file fusion.txt. Questo file viene poi elaborato da pizzly per le analisi successive. (manuale)[https://pachterlab.github.io/kallisto/manual]
8.3 STAR FUSION
Tutorial che usa la potenza di STAR-FUSION tutorial
##. SPLIT FUSION
Le sfide nella diagnosi delle fusioni geniche comprendono: la possibilità di molteplici partner di fusione, talvolta sconosciuti; la bassa espressione genica (ad esempio ALK); l’espressione proteica non specifica per fusione (ad esempio ROS1); il possibile coinvolgimento di siti di splicing criptici; la scarsa diversità di sequenza nei punti di rottura genomici e le conseguenti difficoltà di mappatura; la scarsa qualità o quantità del campione e la bassa cellularità tumorale nei reperti clinici. La tecnologia AMP (Anchored Multiplex PCR) è clinicamente validata e affronta tutte queste problematiche, accelerando le scoperte di fusioni geniche e supportando diagnosi cliniche robuste (Zheng Z, et al. Anchored multiplex PCR for targeted next-generation sequencing. Nat Med. 2014).
Accanto a una tecnologia di laboratorio solida, è altrettanto importante un metodo computazionale ad alte prestazioni per il rilevamento delle fusioni geniche. SplitFusion è rapido grazie all’uso degli allineamenti chimerici split-read di BWA-MEM (Li H. 2013). SplitFusion è indipendente dai trascritti codificanti noti, è sensibile, specifico, computazionalmente efficiente e presenta caratteristiche particolarmente rilevanti per la refertazione clinica, come la capacità di inferire la frame-ness dei trascritti di fusione e gli allineamenti ai confini degli esoni; di calcolare il numero di siti unici di legame DNA/RNA; e la modalità SplitFusion-Target, che consente un miglioramento continuo basato sull’evidenza nella refertazione clinica
SplitFusion può essere utilizzato sia con dati RNA-seq che con dati generati da Anchored Multiplex PCR (AMP).
https://github.com/Zheng-NGS-Lab/SplitFusion
###Come funziona SplitFusion?
L’analisi si articola nei seguenti passaggi computazionali:
Allineamento al riferimento e deduplicazione
Trasformazione CIGAR
Individuazione dei breakpoint candidati
Filtraggio iniziale dei breakpoint
Annotazione dei geni nei breakpoint, determinazione della frame-ness, confini esonici, ulteriori filtraggi e reportistica mirata
Refertazione e visualizzazione dei risultati
8.3.1 RNAFUSION
RNAfusion è una pipeline nf-core per l’analisi di fusioni di RNA-seq.
Fornisce un workflow standardizzato, riproducibile e facilmente estendibile, costruito con Nextflow e containerizzato tramite Docker/Singularity.
9 Caratteristiche principali
- Multipiattaforma e portabile
- Gestione container con Docker/Singularity
- Integrazione con ambienti HPC e cloud
- Report interattivi in HTML
- Test e validazione automatizzati con
nf-core lint
10 Installazione

Per installare ed eseguire la pipeline:
nextflow run nf-core/rnafusion -profile test,docker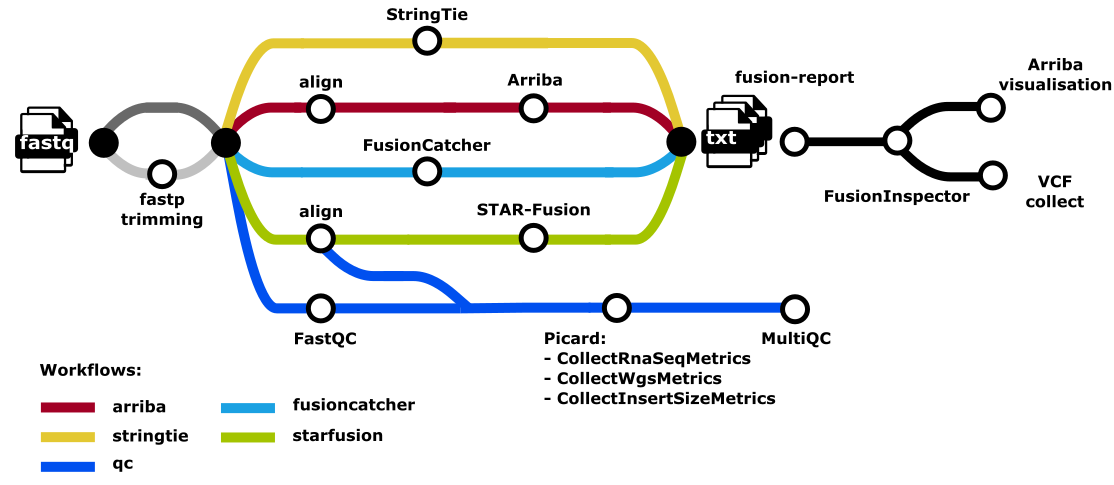
come usarlo è riportato nel seguente link:
https://nf-co.re/rnafusion/3.0.2/docs/usage/
nextflow run nf-core/rnafusion \ --build_references \ --cosmic_username <EMAIL> --cosmic_passwd <PASSWORD> \ --fusionreport \ --genomes_base <PATH/TO/REFERENCES> \ --outdir <OUTPUT/PATH>'''poi fai questo
nextflow run nf-core/rnafusion \
--all \
--input <SAMPLE_SHEET.CSV> \
--genomes_base <PATH/TO/REFERENCES> \
--outdir <OUTPUT/PATH>10.1 Citations
If you use nf-core/rnafusion for your analysis, please cite it using the following doi: 10.5281/zenodo.3946477
An extensive list of references for the tools used by the pipeline can be found in the CITATIONS.md file.
You can cite the nf-core publication as follows:
The nf-core framework for community-curated bioinformatics pipelines.
Philip Ewels, Alexander Peltzer, Sven Fillinger, Harshil Patel, Johannes Alneberg, Andreas Wilm, Maxime Ulysse Garcia, Paolo Di Tommaso & Sven Nahnsen.
Nat Biotechnol. 2020 Feb 13. doi: 10.1038/s41587-020-0439-x.