8 Genefuse
8.1 GeneFuse
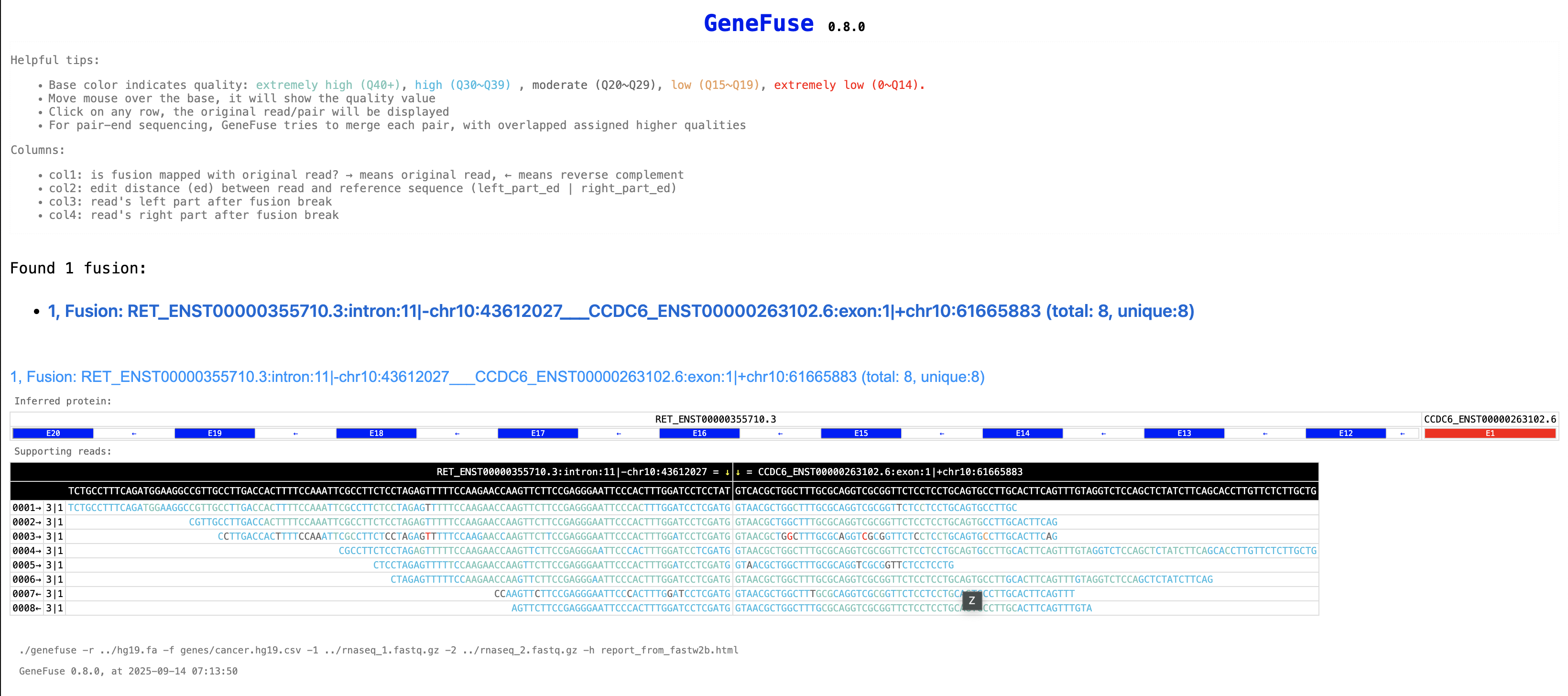
Uno strumento per rilevare e visualizzare fusioni geniche target analizzando direttamente i file FASTQ. Questo tool accetta come input file FASTQ e un genoma di riferimento, e produce come output i risultati delle fusioni rilevate nei formati TEXT, JSON e HTML.
Dai un’occhiata rapida al report informativo
Report HTML di esempio: http://opengene.org/GeneFuse/report.html
Report JSON di esempio: http://opengene.org/GeneFuse/report.json
Dataset per test: http://opengene.org/dataset.html – Scarica i file FASTQ paired-end per testare GeneFuse (piattaforma Illumina).
# Get genefuse program ### install with Bioconda
conda install -c bioconda genefuse8.1.1 download binary
This binary is only for Linux systems, http://opengene.org/GeneFuse/genefuse
# this binary was compiled on CentOS, and tested on CentOS/Ubuntu
wget http://opengene.org/GeneFuse/genefuse
chmod a+x ./genefusesi puo usare sia hg19 e hg38 ## Example
/genefuse -r ../hg19.fa -f genes/druggable.hg19.csv -1 ../rnaseq_1.fastq.gz -2 ../rnaseq_2.fastq.gz -h report_from_fastw2.html > result2a
./genefuse -r ../hg19.fa -f genes/cancer.hg19.csv -1 ../rnaseq_1.fastq.gz -2 ../rnaseq_2.fastq.gz -h report_from_fastw2b.html > result2b
8.1.2 Risultati